No.20104 Re: PLS-DAについて 【青木繁伸】 2013/07/25(Thu) 17:47
使ったことないけど,plsda が返すオブジェクトを str でみると,scores 要素なんでは?> set.seed(4649)
> (x <- matrix(sample(100, 12, replace=TRUE), 4))
[,1] [,2] [,3]
[1,] 96 76 21
[2,] 85 10 48
[3,] 3 71 19
[4,] 73 75 98
> (y <- factor(c(1,1,2,2)))
[1] 1 1 2 2
Levels: 1 2
> ans <- plsda(x, y, ncomp=2, probMethod="Bayes")
> ans$scores
Comp 1 Comp 2
1 -26.72900 -22.19605
2 -38.32675 2.31466
3 45.27848 -35.20478
4 19.77727 55.08616
attr(,"class")
[1] "scores"
scores 関数で取り出すこともできるようだ。> scores(ans)
Comp 1 Comp 2
1 -26.72900 -22.19605
2 -38.32675 2.31466
3 45.27848 -35.20478
4 19.77727 55.08616
attr(,"class")
[1] "scores"
答えが分かっているデータを分析してみると,確証が得られますね。(というか,そうでなければ,安心して使えませんが)
No.20108 Re: PLS-DAについて 【青木繁伸】 2013/07/26(Fri) 18:41
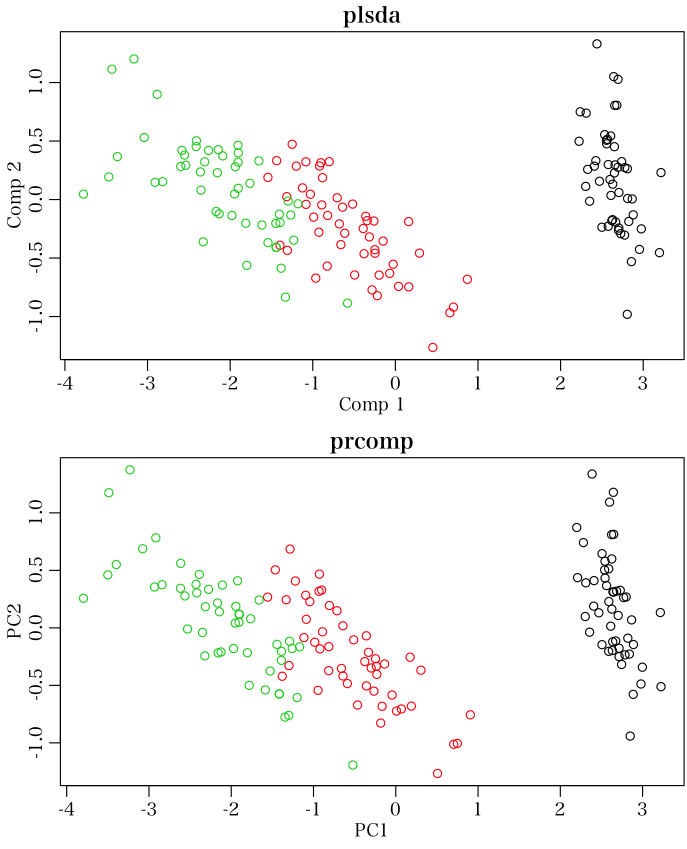
plsda と prcomp で,iris データを分析してみました。
このデータセットでは,第1主成分の寄与率が90%以上もあるので,両者よく似た結果になりますね。
で,scores でよさそうです。ans.plsda <- plsda(iris[, 1:4], iris[,5], ncomp=2, probMethod="Bayes")
ans.prcomp <- prcomp(iris[, 1:4], ncomp=2)
layout(matrix(1:2, 2))
old <- par(mar=c(3, 3, 1.5, 0.5), mgp=c(1.6, 0.6, 0))
plot(scores(ans.plsda), col=rep(1:3, each=50), main="plsda")
plot(-ans.prcomp$x, col=rep(1:3, each=50), main="prcomp")
par(old)